ABSTRAK
Ikan croaker kuning besar adalah salah satu spesies akuakultur laut terpenting di Tiongkok, namun industri pertanian intensifnya menghadapi tantangan dari berbagai patogen, khususnya penyakit bintik putih yang disebabkan oleh Cryptocaryon irritans. Studi ini bertujuan untuk mengatasi masalah penyakit bintik putih melalui pemuliaan genetik. Kami menerapkan dua generasi seleksi genomik (GS) ikan croaker kuning besar berturut-turut terhadap Cryptocaryon irritans , menghasilkan tiga generasi berkelanjutan untuk analisis selanjutnya. Uji tantangan menunjukkan tingkat kelangsungan hidup 96 jam yang jauh lebih tinggi pada generasi yang dipilih dibandingkan dengan kontrol yang sesuai, dengan peningkatan masing-masing sebesar 18,5% dan 79,7%. Analisis kelangsungan hidup mengonfirmasi bahwa dua generasi yang dipilih menunjukkan ketahanan yang jauh lebih kuat terhadap C. irritans . Dengan menggabungkan file genotipe lintas generasi, kumpulan data komprehensif yang berisi 1844 individu dan 28.637 SNP dibuat. Nilai Pemuliaan Diperkirakan Genomik (GEBV) menunjukkan peningkatan yang stabil di tiga generasi berturut-turut, sementara analisis struktur genetik mengungkapkan diferensiasi populasi progresif yang dihasilkan dari dua putaran GS. Melalui pemindaian tanda seleksi genom secara luas, kami mengidentifikasi lima daerah seleksi positif (PSR) yang didistribusikan melintasi empat kromosom. Daerah-daerah ini diperkaya untuk beberapa jalur biologis yang terkait dengan metabolisme energi, respons imun, dan kematian sel, termasuk jalur pensinyalan HIF-1, jalur pensinyalan reseptor mirip NOD, dan apoptosis. Dalam jalur-jalur ini, kami mengidentifikasi gen kandidat utama, termasuk crebbp dalam jalur pensinyalan HIF-1 dan traf2 yang terlibat dalam regulasi imun, keduanya secara signifikan terkait dengan resistensi terhadap C. irritans . Hasil kami memvalidasi efektivitas GS dalam pembiakan selektif ikan croaker kuning besar terhadap C. irritans dan menunjukkan bahwa hanya dua generasi GS berturut-turut dapat menginduksi diferensiasi substansial dalam struktur genetik. Pendekatan ini memfasilitasi identifikasi gen kandidat dan jalur biologis yang terkait dengan resistensi penyakit.
1 Pendahuluan
Ikan croaker kuning besar ( Larimichthys crocea ) adalah salah satu spesies akuakultur laut yang paling penting secara ekonomi di Tiongkok, dengan sejarah panjang signifikansi budaya dalam makanan. Karena penangkapan ikan yang berlebihan selama tahun 1950-an dan 1980-an, sumber daya perikanan ikan croaker kuning besar telah menipis (Liu dan de Mitcheson 2008 ). Setelah terobosan reproduksi buatan, produksi tahunan sekarang terutama berasal dari akuakultur. Namun, industri akuakultur intensif yang terletak di dalam fjord sering kali terganggu oleh wabah berbagai organisme patogen, yang menyebabkan kerugian ekonomi yang signifikan. Penyakit yang paling parah adalah penyakit bercak putih, yang disebabkan oleh parasit ciliata Cryptocaryon irritans . Pada suhu musim panas yang sesuai, siklus hidup C. irritans dipersingkat dan sering kali menyebabkan infeksi berlebihan, yang akhirnya menyebabkan kematian ikan (Li et al. 2022 ). Dibandingkan dengan spesies lain, ikan kerapu kuning besar lebih rentan terhadap infeksi C. irritans (Yin et al. 2018 ). Ada kebutuhan mendesak untuk mengurangi kerugian yang disebabkan oleh penyakit bercak putih pada industri budidaya ikan kerapu kuning besar. Mengingat kurangnya tindakan yang hemat biaya dan hemat tenaga kerja untuk pencegahan dan pengendalian penyakit, memanfaatkan seleksi genetik untuk mengembangkan galur baru ikan kerapu kuning besar dengan ketahanan yang lebih baik terhadap penyakit bercak putih merupakan solusi yang paling menjanjikan.
Pengembangan seleksi buatan memiliki sejarah jangka panjang, dari domestikasi di zaman kuno hingga pembiakan berbantuan molekuler modern. Sepanjang sejarah pembiakan selektif, fokus telah bergeser dari sifat Mendelian sederhana ke sifat kuantitatif yang lebih kompleks. Selain itu, seleksi individu unggul telah berevolusi dari metode berbasis fenotipe ke pendekatan berbasis nilai pembiakan. Estimasi nilai pembiakan juga telah beralih dari yang hanya didasarkan pada informasi silsilah menjadi penggabungan data penanda molekuler yang ekstensif. Saat ini, seleksi genomik (GS) adalah salah satu teknologi pembiakan genetik paling mutakhir. GS adalah alat pembiakan yang kuat yang memanfaatkan data genomik untuk memprediksi nilai pembiakan individu berdasarkan genotipe mereka. Metode ini mengintegrasikan informasi genomik ke dalam proses pembiakan dengan memperkirakan efek dari ribuan penanda genetik yang didistribusikan di seluruh genom. Estimasi ini digunakan untuk memprediksi kinerja individu yang belum diuji, memungkinkan peningkatan genetik yang lebih akurat dan lebih cepat. Sejak diperkenalkan oleh Dr. Meuwissen Theo (Meuwissen et al. 2001 ), GS telah berevolusi secara bertahap dari sebuah konsep teoritis ke sebuah aplikasi praktis, didorong oleh kemajuan dalam teknologi sekuensing berthroughput tinggi. Saat ini, GS telah digunakan secara luas dalam pemuliaan sifat-sifat kompleks ternak, tanaman pangan, dan hewan akuatik, dan telah memperoleh prestasi yang luar biasa. Misalnya, seleksi genom telah merevolusi pembiakan sapi perah di Amerika Serikat. Dalam kurun waktu tujuh tahun setelah penerapan GS pada tahun 2009, tingkat perolehan genetik pada ras Holstein per tahun meningkat dari ~50% menjadi 100% untuk sifat-sifat hasil (susu, lemak, protein) dan dari tiga kali lipat menjadi empat kali lipat untuk sifat-sifat yang dapat diwariskan rendah (fertilitas sapi, masa hidup dalam kawanan, kesehatan kelenjar susu) (García-Ruiz et al. 2016 ). Kini, produsen telah menerima evaluasi genomik sebagai indikasi akurat potensi seekor pejantan, menggantikan evaluasi berbasis anakan tradisional (Wiggans et al. 2017 ). Dalam hal penerapan GS pada hewan akuatik, keandalan seleksi genomik pertama kali dibahas menggunakan validasi silang dalam pembiakan salmon Atlantik untuk ketahanan terhadap kutu laut pada tahun 2014, dan hasilnya mengungkapkan bahwa model genomik mengungguli model berbasis silsilah klasik (Odegård et al. 2014 ). Selanjutnya, kelayakan teoritis GS juga ditunjukkan dalam pembiakan hewan akuatik lainnya (Houston et al. 2020 ; Yáñez et al. 2023 ). Perlu dicatat bahwa dalam pengembangbiakan ikan croaker kuning besar, salmon Atlantik, dan ikan trout pelangi, para peneliti telah menggunakan pengujian keturunan untuk lebih mengkonfirmasi kepraktisan GS (Vallejo et al. 2016 , 2024 ; Verbyla et al. 2022).; Zhao, Bai, dkk. 2021 ). Kasus-kasus yang berhasil ini telah sangat mendorong penggunaan GS dalam pemuliaan hewan akuatik. Selain itu, galur-galur yang diperoleh dengan cepat menggunakan GS akan menjadi bahan percobaan yang berharga untuk analisis genetik sifat-sifat target, seperti pemindaian tanda-tanda seleksi di seluruh genom yang terkait dengan sifat-sifat tersebut.
Tanda-tanda seleksi didefinisikan sebagai daerah genom yang mengandung urutan DNA yang terlibat dalam variasi genetik sifat-sifat yang mengalami seleksi alam atau buatan (López et al. 2015 ; Qanbari et al. 2012 ). Tanda-tanda seleksi dapat muncul sebagai akibat dari efek hitchhiking, di mana lokus genetik yang terkait dengan alel yang menguntungkan meningkat frekuensinya karena kedekatan fisiknya dengan target seleksi yang sebenarnya (Smith dan Haigh 2007 ). Namun, penting untuk menyadari bahwa beberapa lokus sendiri merupakan target langsung seleksi, dan perubahan frekuensinya merupakan akibat langsung dari seleksi yang bekerja pada sifat yang diinginkan, bukan karena keterkaitan dengan situs terpilih lainnya. Karena kemajuan dalam teknologi genom dan pengurangan biaya genotipe, penelitian yang terkait dengan pemindaian genom tanda-tanda seleksi pada hewan akuatik telah meningkat dalam beberapa tahun terakhir. Tren ini dimotivasi oleh keberhasilan mengaitkan gen yang terlibat dalam tanda-tanda seleksi dengan adaptasi lingkungan atau sifat-sifat menguntungkan lainnya. Misalnya, penelitian tentang pemindaian tanda-tanda seleksi untuk populasi ikan croaker kuning besar liar telah mengungkapkan bahwa gen yang terkait dengan proses perkembangan berada di bawah seleksi alam yang kuat pada populasi utara, sedangkan gen yang terkait dengan kekebalan ditemukan dipilih pada populasi selatan (Chen, Bai, et al. 2023 ). Sejumlah penelitian pada spesies akuatik lainnya juga menunjukkan bahwa gen fungsional telah menjadi subjek seleksi yang kuat dalam proses adaptasi yang panjang terhadap lingkungan yang beragam (Chen, Zhou, et al. 2023 ; Shen et al. 2019 ). Karena sebagian besar spesies ikan memiliki sejarah domestikasi yang pendek, banyak penelitian telah difokuskan pada pendeteksian tanda-tanda seleksi yang relatif baru. Misalnya, sebuah penelitian yang mencari tanda-tanda seleksi pada populasi salmon Atlantik Cermaq yang dibudidayakan, dengan sejarah pembiakan selektif sekitar 50 tahun, mengungkapkan bahwa gen dengan fungsi molekuler yang berpotensi terkait dengan pertumbuhan, respons terhadap patogen dan stresor lingkungan, dan pematangan seksual awal berada di bawah seleksi yang kuat (Gutierrez et al. 2016 ). Studi serupa lainnya pada ikan trout pelangi yang dibudidayakan juga menemukan bahwa gen kandidat yang terlibat dalam tanda seleksi dikaitkan dengan sifat-sifat seperti pertumbuhan, perkembangan awal, reproduksi, perilaku, dan fungsi sistem kekebalan tubuh (Cádiz et al. 2021) .). Literatur terbatas tersedia untuk mendeteksi tanda seleksi pada tahap awal seleksi buatan dan menyelidiki dampak seleksi buatan jangka pendek pada struktur populasi, bahkan ketika seleksi telah diterapkan hanya untuk satu atau beberapa generasi. Karena GS memberikan akurasi estimasi nilai pembiakan yang lebih tinggi daripada metode berbasis silsilah tradisional, metode ini dapat mempercepat perbaikan genetik. Oleh karena itu, sangat penting untuk menilai dampak GS dengan mendeteksi apakah GS jangka pendek dapat menyebabkan tanda seleksi dan menyebabkan diferensiasi genetik.
Di sini, kami menggunakan dua praktik GS untuk menghasilkan dua generasi berturut-turut galur yang resistan terhadap C. irritans dalam pembiakan selektif resistensi bercak putih pada ikan kerapu kuning besar. Kami kemudian mengevaluasi divergensi struktur populasi skala halus di tiga generasi berturut-turut dan mendeteksi tanda-tanda seleksi di seluruh genom selama kedua siklus GS untuk menyelidiki mekanisme yang mendasari resistensi bercak putih. Hal yang menonjol dari studi kami adalah bahwa tanda-tanda genomik seleksi dijelaskan melalui analisis garis keturunan yang mengalami pembiakan yang dipercepat melalui protokol GS, melengkapi pendekatan Studi Asosiasi Genom-Lebar (GWAS) konvensional kami. Desain eksperimental ini memungkinkan interogasi arsitektur genetik yang mendasari fenotipe resistan melalui dua metodologi: mengidentifikasi daerah genomik yang menunjukkan jejak seleksi, sementara juga memeriksa asosiasi langsung dengan varians fenotipik. Metodologi berbasis seleksi kami memberikan wawasan pelengkap untuk studi asosiasi tradisional dengan menangkap lokus yang responsif terhadap tekanan seleksi buatan, yang berpotensi mengungkap elemen genetik fungsional yang mungkin tidak terlihat dalam GWAS standar karena interaksi genetik yang kompleks atau ketergantungan lingkungan. Pendekatan terpadu ini menawarkan pemahaman yang lebih komprehensif tentang dasar genetis untuk sifat target daripada yang dapat diberikan oleh salah satu metode saja.
2 Bahan dan Metode
2.1 Persetujuan Etis
Penelitian ini disetujui oleh Komite Etika dan Manajemen Hewan Laboratorium, Fakultas Ilmu Kelautan dan Bumi, Universitas Xiamen. Semua prosedur eksperimen yang melibatkan ikan kerapu kuning besar dilakukan sesuai dengan Peraturan untuk Administrasi Urusan Mengenai Hewan Eksperimen (Fakultas Ilmu Kelautan dan Bumi, Universitas Xiamen).
2.2 Metodologi Seleksi Genom
Prosedur untuk menerapkan Seleksi Genomik (GS) dijelaskan secara rinci dalam studi kami sebelumnya (Zhao, Bai, et al. 2021 ). Singkatnya, proses GS terdiri dari dua langkah utama. Langkah pertama melibatkan pembentukan populasi referensi, yang untuknya data fenotipik dan genotipik diperoleh. File genotipe biasanya mencakup ribuan lokus genetik, dan hubungan regresi matematis antara fenotipe dan genotipe dikembangkan untuk membentuk model seleksi genomik. Langkah kedua memerlukan pembuatan populasi kandidat seleksi, mengurutkan genom mereka untuk mendapatkan genotipe mereka, dan menggunakan model seleksi genomik untuk menghitung nilai pemuliaan genomik (GEBV) untuk kandidat. Berdasarkan GEBV, individu unggul dapat dipilih untuk mereproduksi generasi berikutnya.
Dalam studi ini, GS diimplementasikan menggunakan paket BGLR (Bayesian Generalized Linear Regression) di R. BGLR adalah kerangka kerja yang fleksibel untuk mengimplementasikan berbagai model Bayesian, yang khususnya berguna untuk seleksi genomik dalam sifat-sifat yang kompleks. Data genomik, yang biasanya terdiri dari genotipe SNP, dikodekan dalam format matriks, di mana baris-barisnya sesuai dengan individu dan kolom-kolomnya mewakili SNP. Data fenotipe (pengukuran sifat) untuk setiap individu juga disusun dalam format vektor. Model prediksi genomik dasar dinyatakan sebagai:
![]()
di mana y adalah vektor nilai fenotipik, X adalah matriks genotipe, β merupakan vektor efek penanda, dan ϵ adalah vektor residual. Dalam studi ini, model Bayesian LASSO digunakan, di mana prior untuk efek SNP ditetapkan menggunakan distribusi Laplace. Dengan menggunakan MCMC, distribusi posterior efek penanda β diestimasi. BGLR menggunakan algoritma seperti pengambilan sampel Gibbs untuk memperbarui nilai parameter secara berulang, termasuk efek genetik, hingga konvergensi tercapai. Dalam studi ini, 50.000 iterasi ditetapkan. Proses iteratif ini memungkinkan model untuk menghitung rerata posterior SNP, yang selanjutnya digunakan untuk memprediksi nilai genetik individu yang belum diuji. Setelah distribusi posterior efek SNP diperoleh, GEBV untuk setiap individu dalam populasi kandidat seleksi dapat diprediksi menggunakan rumus:
![]()
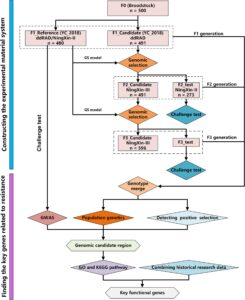
2.3 Tiga Generasi Ikan Croaker Kuning Besar yang Berturut-turut
Dua GS berturut-turut diaplikasikan untuk meningkatkan kinerja resistensi ikan croaker kuning besar terhadap C. irritans , sehingga menghasilkan dua galur resisten berturut-turut. Kedua generasi resisten ini (F2 dan F3), bersama dengan generasi pertama (F1), yang tidak menjalani seleksi untuk resistensi C. irritans , menghasilkan tiga generasi berturut-turut untuk analisis berikut. Dalam studi saat ini, populasi referensi yang sama digunakan seperti dalam studi GS sebelumnya (Zhao, Bai, et al. 2021 ). Secara singkat, populasi referensi diproduksi pada bulan Januari 2018, yang terdiri dari keturunan 500 induk (generasi F0) dari populasi geografis lokal di Ningde, Tiongkok. Infeksi buatan C. irritans pada populasi referensi dilakukan pada bulan Juli 2018 untuk mendapatkan fenotipe resisten, yang diwakili oleh waktu kematian dan status kelangsungan hidup. Karena sifat biner (status bertahan hidup) lebih dekat dengan sifat resistensi penyakit daripada sifat kuantitatif (waktu kematian), kami menggunakan status bertahan hidup dalam praktik GS. GS pertama diterapkan pada bulan Februari 2020. Kandidat yang dipilih berasal dari populasi yang sama dengan populasi referensi, dan kedua populasi ini diperlakukan sebagai generasi F1. Sebanyak 50 individu dengan GEBV tinggi dipilih dari kandidat seleksi F1 ( n = 491) untuk mereproduksi generasi berikutnya, F2, yang secara teoritis diharapkan menunjukkan kinerja yang baik terhadap C. irritans . Ketika generasi F2 mencapai usia empat bulan, yang bertepatan dengan seringnya terjadinya penyakit bercak putih, uji tantangan C. irritans dilakukan untuk menilai kinerja resistensi. Dua tahun kemudian, kami menerapkan praktik GS kedua. Populasi referensi, sama seperti yang digunakan dalam GS pertama, digunakan untuk menghitung GEBV untuk kandidat seleksi F2 ( n = 523). Secara total, 50 individu dengan GEBV tinggi dipilih sebagai indukan untuk mereproduksi generasi F3. Uji tantang untuk generasi F3 dilakukan pada bulan Juni 2022.
2.4 Pengurutan dan Genotipe
Pendekatan sekuensing DNA terkait restriksi pencernaan ganda (ddRAD) digunakan dalam GS pertama. Dari populasi referensi F1, 480 individu, termasuk 240 kematian dan 240 yang bertahan hidup, dan 499 individu dalam kandidat seleksi F1 dipilih untuk mengekstrak DNA genomik dan membangun pustaka ddRAD, yang dikirim ke Novogene Technology Co. (Tianjin, Tiongkok) untuk sekuensing berpasangan Illumina HiSeq 150 bp. Program STACKS v2 dan BWA v0.7.17 digunakan untuk memetakan pembacaan bersih ke genom referensi, yang memungkinkan akuisisi genotipe mentah. Kontrol kualitas untuk SNP diimplementasikan menggunakan program PLINK v1.9, dengan tingkat pemanggilan individu lebih tinggi dari 0,90, tingkat pemanggilan lokus SNP lebih tinggi dari 0,9, dan frekuensi alel minor lebih tinggi dari 0,05. Program BEAGLE v5.0 dan HAPLOVIEW v4.2 digunakan untuk menghitung genotipe yang hilang dan menemukan SNP penandaan. Setelah penyaringan, berkas genotipe dengan 942 individu dan 12.385 SNP dibuat untuk praktik GS pertama.
Dalam praktik GS kedua, strategi sekuensing diperbarui menjadi sekuensing chip, yang memastikan stabilitas dan konsistensi lokus SNP. Populasi referensi F1 yang sama yang digunakan dalam GS pertama juga diperlakukan sebagai populasi referensi untuk GS kedua. Perbedaannya adalah bahwa populasi referensi digenotipe dengan array fase padat Large Yellow Croaker 55 K NingXin-II (Zhou et al. 2022 ). Kandidat seleksi F2 digenotipe menggunakan array fase cair Large Yellow Croaker 55 K NingXin-III, yang berisi panel SNP yang hampir sama dengan array fase padat NingXin-II (Wang et al. 2023 ). Alasan menggunakan array NingXin-III adalah karena lebih hemat biaya daripada array NingXin-II. Genotipe populasi referensi F1 dan kandidat seleksi F2 digabungkan menjadi satu berkas genotipe dan diikuti oleh kontrol kualitas, dengan tingkat pemanggilan lokus SNP lebih tinggi dari 0,99 dan frekuensi alel minor lebih tinggi dari 0,05. Berkas genotipe yang telah dibersihkan yang berisi 1003 individu dan 41.551 SNP digunakan dalam praktik GS kedua.
Subset individu ( n = 273) dalam generasi F2, yang telah diuji untuk tantangan C. irritans , juga digenotipe menggunakan array NingXin-II. Sebanyak 30 individu yang diambil sampelnya secara acak dari generasi F3 berumur satu tahun dan kandidat seleksi F3 digenotipe menggunakan array NingXin-III. Populasi yang digunakan dalam berbagai generasi dirangkum dalam tabel di bawah ini (Tabel 1 ). Data genotipe chip untuk berbagai generasi digabungkan menjadi satu berkas genotipe. Data ddRAD tidak disertakan karena data chip dan ddRAD memiliki lebih sedikit lokus SNP yang sama. Setelah kontrol kualitas data genotipe chip, 1844 individu dan 28.637 SNP dipertahankan untuk analisis struktur populasi dan pemindaian genom tanda-tanda seleksi yang terkait dengan fenotipe resistan.
| Populasi | Metode pengurutan | Jumlah individu |
|---|---|---|
| Populasi referensi F1 | susunan ddRAD/NingXin-II | 480 |
| Kandidat seleksi F1 | ddRAD | 491 |
| uji F2 | Susunan NingXin-II | 273 |
| Kandidat seleksi F2 | Susunan NingXin-III | 523 |
| F3 dipilih secara acak | Susunan NingXin-III | 30 |
| Kandidat seleksi F3 | Susunan NingXin-III | 566 |
2.5 Uji Tantangan C. irritans
Siklus hidup C. irritans terdiri dari tiga tahap utama: trophont, tomont, dan theront. Trophont adalah tahap parasit yang berada di dalam epitel inang, memberi makan dan menyebabkan kerusakan jaringan. Setelah matang, ia terlepas dan membentuk tomont, tahap reproduksi berkista di lingkungan eksternal tempat banyak theront diproduksi. Theront adalah tahap infeksius yang berenang bebas yang bertanggung jawab untuk memulai infeksi baru. Prosedur untuk tantangan C. irritans dijelaskan dalam studi GS kami sebelumnya (Zhao, Bai, et al. 2021 ). Secara singkat, tomont yang terbentuk dari trophont yang menempel pada inang (ikan croaker kuning besar dalam penelitian ini) dikumpulkan dari dasar tangki. Setelah sekitar dua hari inkubasi tomont pada suhu 26°C, theront dilepaskan. Akhirnya, jumlah theront dalam 10 μL air laut diperkirakan dengan pengambilan sampel acak berganda untuk melakukan infeksi kuantitatif. Tantangan C. irritans diterapkan untuk generasi F2 dan F3 pada usia sekitar empat bulan untuk menguji apakah keturunannya menunjukkan kinerja resistensi yang lebih baik. Galur komersial, yang diperbanyak dan dipelihara selama periode yang sama dengan generasi F2 dan F3, diperlakukan sebagai kelompok kontrol dalam uji tantangan. Tiga replikasi kelompok eksperimen dan kontrol digunakan, dengan masing-masing berisi 150 individu yang ditempatkan dalam tangki baja plastik (volume = 1 m 3 ). Dosis tantangan ditentukan dari uji coba percontohan dan ditetapkan pada 2000 dan 2500 theront per ikan dalam uji tantangan F2 dan F3, masing-masing. Dosis tantangan yang berbeda yang digunakan untuk generasi yang berbeda dapat dikaitkan dengan berbagai faktor, seperti ukuran ikan percobaan dan infektivitas theront. Setelah infeksi, pengamatan berlangsung selama 96 jam, dan waktu kematian setiap individu dicatat. Kriteria untuk menentukan kematian adalah ikan percobaan kehilangan keseimbangan dan tidak dapat kembali ke kondisi berenang normal dalam waktu 10 detik setelah disentuh. Paket survival dalam R versi 4.2.0 digunakan untuk melakukan analisis survival pada kelompok percobaan dan kontrol.
2.6 Analisis Struktur Populasi
Statistik dasar dihitung, termasuk heterozigositas yang diamati ( H o), heterozigositas yang diharapkan ( H e), dan koefisien perkawinan sedarah ( F h dan F roh) untuk setiap generasi menggunakan program PLINK v1.9. Nilai F h adalah estimasi kanonik F genomik berdasarkan homozigositas SNP berlebih, sedangkan nilai Froh mengacu pada proporsi genom yang berada dalam rangkaian homozigositas. Keragaman nukleotida ( π ) pada setiap kromosom dihitung menggunakan VCFtools v0.1.17. Kami menggunakan tiga metode untuk mengungkap struktur genetik dari berbagai generasi: analisis komponen utama (PCA), analisis komponen leluhur, dan analisis pohon. PCA dilakukan menggunakan program PLINK v1.9. Program ADMIXTURE v1.3.0 digunakan untuk memperkirakan leluhur genetik setiap sampel, dan paket pophelper di R v4.2.0 digunakan untuk memplot hasilnya. Kami menggunakan program VCF-kit v0.2.6 untuk mengimplementasikan analisis pohon berdasarkan format VCF dan paket ggtree di R v4.2.0 untuk menampilkan hasilnya.
2.7 Mendeteksi Tanda Genom Seleksi
Kami menggunakan berbagai metode untuk mengidentifikasi seleksi positif dan mendeteksi tanda-tanda seleksi selama dua GS berturut-turut, termasuk indeks fiksasi (Fst), perubahan lipatan dalam keanekaragaman nukleotida ( ω ), dan tingkat homozigositas haplotipe (Rsb). Indeks-indeks ini diperkirakan pada jendela genomik geser 100 kb dengan langkah 10 kb. Nilai Fst dan keanekaragaman nukleotida ( π ) untuk setiap jendela geser dihitung menggunakan VCFtools v0.1.17. Logaritma rasio nilai π untuk dua populasi dihitung sebagai indeks ω . Nilai Rsb dihitung untuk setiap SNP menggunakan paket rehh di R v4.2.0 dan kemudian dirata-ratakan di setiap jendela geser. Dua langkah diambil untuk mengidentifikasi daerah yang dipilih secara positif (PSR).
Pertama, indeks skor seleksi komposit (CSS) dihitung berdasarkan Fst, ω , Rsb, dan nilai- p mengikuti metode yang dijelaskan sebelumnya (Avalos et al. 2017 ). Nilai- p diperoleh dari analisis GWAS untuk populasi referensi F1. Prosedur analisis GWAS dilakukan menurut studi GWAS kami sebelumnya (Zhao, Zhou, et al. 2021 ). Karena indeks ω dan Rsb dihitung berdasarkan dua populasi, kami menggunakan persamaan berikut untuk menghitung kedua indeks ini berdasarkan tiga generasi dalam studi saat ini, yang mencerminkan fluktuasi total di seluruh proses dua seleksi genetik:

3 Hasil
3.1 Kinerja Ketahanan Terhadap C. irritans pada Generasi F2 dan F3
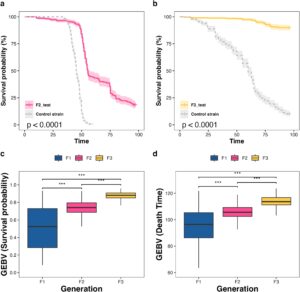
Untuk mengonfirmasi efektivitas GS, uji tantangan C. irritans dilakukan pada generasi F2 dan F3 untuk mendeteksi apakah resistensinya telah ditingkatkan. Hasil uji tantangan untuk generasi F2 menunjukkan bahwa tingkat kelangsungan hidup 96 jam dari generasi F2 adalah 18,5%, sedangkan kelompok kontrol menurun menjadi 0 (Gambar 2a ). Hasil uji tantangan untuk generasi F3 menunjukkan bahwa tingkat kelangsungan hidup 96 jam adalah 89,9%, sedangkan kelompok kontrol hanya 10,2% (Gambar 2b ). Analisis kelangsungan hidup mengungkapkan bahwa dibandingkan dengan kelompok kontrol, kinerja resistensi generasi F2 dan F3 ditingkatkan secara signifikan, yang menunjukkan efektivitas dan kepraktisan teknologi GS dalam seleksi genetik sifat-sifat kompleks. Perlu dicatat bahwa karena kerentanan uji tantangan terhadap faktor lingkungan eksternal, perbandingan yang berarti antara hasil tantangan di berbagai generasi tidak mungkin dilakukan. Lebih jauh, signifikansi kualitatif dari uji tantangan lebih besar daripada signifikansi kuantitatifnya. Oleh karena itu, kami menghitung GEBV dari tiga generasi, karena GEBV memberikan indikasi yang lebih baik tentang peningkatan resistensi pada setiap generasi. Hasilnya menunjukkan bahwa terlepas dari apakah fenotipe resistensi diukur berdasarkan waktu kematian atau status kelangsungan hidup, GEBV meningkat pada setiap generasi (Gambar 2c,d ), yang secara tidak langsung menunjukkan efektivitas dua generasi GS yang berurutan.
3.2 Diferensiasi Struktur Populasi dalam Tiga Generasi Berturut-turut
Parameter populasi dasar diestimasi untuk menentukan status setiap generasi (Tabel 2 ). Untuk heterozigositas yang diamati ( H o) dan koefisien perkawinan sedarah berdasarkan homozigositas SNP ( F h), tidak ada perbedaan signifikan di antara ketiga generasi, tetapi koefisien perkawinan sedarah berdasarkan ROH meningkat seiring dengan generasi (Gambar S1a ), yang mengindikasikan bahwa koefisien perkawinan sedarah menunjukkan tren peningkatan dengan pembiakan genetik berkelanjutan. Heterozigositas yang diharapkan ( H e) dan keanekaragaman nukleotida ( π ) keduanya menurun seiring dengan generasi (Gambar S1b ), tetapi tidak mencapai tingkat signifikan, yang menunjukkan bahwa seiring berlanjutnya seleksi, keanekaragaman genetik menunjukkan tren penurunan.
| Generasi | H atau | Dia | F jam | F roh | π |
|---|---|---|---|---|---|
| Bahasa Indonesia: F1 | 0.288 | 0.300 | 0,025 | 0,0534 tahun | Nomor 8.969E−06 |
| F2 | 0.296 | 0.292 | -0,003 | 0,0587 tahun | Nomor 8.719E−06 |
| F3 | 0.293 | 0,277 tahun | 0,006 | 0,0748 pukul 0,0748 | Nomor 8.278E−06 |
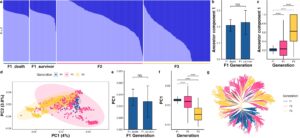
Kemudian struktur genetik setiap generasi diselidiki melalui beberapa metode. Plot skor PCA dua dimensi mengungkapkan bahwa tidak ada struktur pengelompokan independen yang signifikan di antara ketiga generasi (Gambar 3d ). Namun, kami menemukan bahwa seiring bertambahnya generasi, struktur kelompok secara bertahap bergeser ke kiri; dengan kata lain, nilai rata-rata komponen utama pertama (PC1) secara bertahap menurun (Gambar 3e ), dan nilai PC1 rata-rata yang selamat dalam populasi referensi F1 lebih rendah daripada individu yang mati (Gambar 3f ), yang menunjukkan bahwa seleksi menuju ke arah yang diharapkan dan diinginkan. Analisis komponen leluhur juga mengonfirmasi hasil di atas. Ketika K = 2, nilai rata-rata komponen leluhur 1 meningkat seiring dengan generasi (Gambar 3a ), dan nilai rata-rata komponen leluhur 1 ini dari yang selamat dalam populasi referensi F1 lebih tinggi daripada individu yang mati (Gambar 3b ). Pohon filogenetik juga mendukung struktur populasi yang jelas, bertepatan dengan hasil PCA dan analisis komponen leluhur (Gambar 3g ). Generasi F1 dan F3 memiliki struktur topologi yang relatif independen dalam plot pohon, dan kelompok generasi F2 tercampur di antara keduanya. Singkatnya, struktur genetik dari tiga generasi berturut-turut dengan jelas mengungkapkan keberadaan dan arah seleksi buatan dan menunjukkan kekuatan GS.
3.3 Daerah yang Dipilih Secara Positif di Bawah Dua GS Berturut-turut
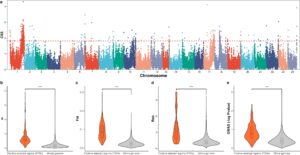
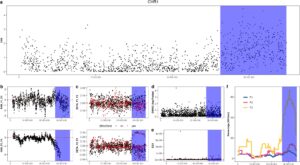
Analisis GWAS pada populasi referensi F1 diimplementasikan, dan tiga indeks ( ω , Fst, dan Rsb) digunakan untuk memindai tanda tangan seleksi genomik (Gambar S2 ). Kami kemudian menggunakan nilai CSS yang terintegrasi dari keempat indeks di atas untuk menyaring PSR awal (Gambar 4a ). Sebanyak 19 PSR awal terdeteksi, dan PSR ini ditemukan terdistribusi pada 11 kromosom (Tabel S1 ). Nilai rata-rata dari empat indeks independen dalam PSR awal secara signifikan lebih tinggi daripada di seluruh genom (Gambar 4b–e ), yang menunjukkan bahwa indeks CSS memiliki representasi yang lebih baik untuk mendeteksi tanda tangan seleksi. Setelah penyaringan lebih lanjut berdasarkan empat kriteria, lima PSR yang terletak pada kromosom 1, 10, 17, dan 18 dipertahankan (Tabel 3 ). Dalam dua GS, tingkat homozigositas haplotipe dalam PSR pada kromosom 1 meluas (Gambar 5b ). Dalam PSR ini, jumlah SNP dengan pergeseran alel pada arah yang diinginkan ( n = 188) jauh melebihi seperempat dari semua SNP (Gambar 5c ). Selain itu, nilai- p GWAS dan EGV dalam PSR ini lebih tinggi daripada di wilayah sekitarnya (Gambar 5d,e ). Selain itu, frekuensi ROH meningkat seiring dengan generasi, yang menunjukkan bahwa PSR ini berada di bawah seleksi yang kuat (Gambar 5f ). Informasi PSR lainnya mirip dengan deskripsi di atas, yang disediakan dalam plot tambahan (Gambar S3– S5 ).
| Krom | Bin_mulai | Bin_akhir | N_varian | N_arah_kanan |
|---|---|---|---|---|
| 1 | 26.250.001 | 34.872.296 | 367 | 188 |
| 10 | 19.770.001 | 21.420.000 | 83 | 48 |
| 17 | 14.680.001 | 16.850.000 | 102 | 47 |
| 17 | 21.580.001 dolar AS | 24.960.000 | 166 | 87 |
| 18 | 13.170.001 dolar AS | 17.440.000 | 208 | 107 |
3.4 Jalur Fungsional dan Gen di Daerah Terpilih Positif Terkait dengan Resistensi terhadap C. irritans
Sebanyak 750 gen diidentifikasi dalam PSR. Analisis pengayaan GO dan KEGG dilakukan untuk gen-gen ini. Semua istilah GO yang diberi anotasi dan jalur KEGG tercantum dalam tabel suplementer (Tabel S2 dan S3 ), dan 20 istilah GO dan jalur KEGG teratas dengan faktor pengayaan tinggi disediakan dalam gambar suplementer (Gambar S6 dan S7 ). Menggabungkan data transkriptomik dan GWAS yang dipublikasikan sebelumnya tentang infeksi C. irritans pada ikan croaker kuning besar, banyak jalur biologis yang terlibat dalam respons molekuler terhadap infeksi C. irritans juga diidentifikasi dalam PSR yang terdeteksi dalam penelitian ini. Jalur biologis penting ini terkait dengan metabolisme energi dan respons imun (Gambar 6a ). Jalur biologis yang terkait dengan metabolisme energi mencakup “jalur pensinyalan Apelin,” “jalur pensinyalan Fosfolipase D,” “jalur pensinyalan Glukagon,” “Degradasi asam lemak,” “jalur pensinyalan HIF-1,” dan lainnya. Jalur biologis yang terkait dengan respons imun meliputi “jalur pensinyalan reseptor mirip NOD,” “pengaturan mediator inflamasi pada saluran TRP,” dan “jalur pensinyalan reseptor sel B.” Ada pula jalur yang terkait dengan kematian sel, seperti “Autofagi” dan “Apoptosis.”

Dalam penelitian sebelumnya tentang respons molekuler ikan croaker kuning besar terhadap infeksi C. irritans , jalur pensinyalan HIF-1 sering disorot (Bai, Wang, Zhao, Bai, et al. 2022 ; Zhang et al. 2020 ), karena parasitisme theronts menghancurkan jaringan insang, menyebabkan keadaan hipoksia, dan dengan demikian menginduksi respons jalur pensinyalan HIF-1. Dalam penelitian saat ini, kami menemukan bahwa crebbp (protein pengikat Creb, Creb adalah protein pengikat elemen respons cAMP), gen yang terlibat dalam jalur pensinyalan HIF-1, berada di bawah seleksi kuat. Gen crebbp terletak di PSR pada kromosom 1. Ada SNP (LG1_31085536) di intron pertama crebbp (Gambar 6b ). Frekuensi alel SNP ini pada yang bertahan hidup dan yang mati pada populasi referensi F1 masing-masing adalah 0,361 dan 0,265 (Gambar 6c ), dan berubah menjadi 0,368 pada generasi F2, dan akhirnya meningkat menjadi 0,772 pada generasi F3 (Gambar 6d ), yang menunjukkan bahwa SNP ini cenderung menjadi tetap dalam tiga generasi. Selain itu, gen kandidat kunci lainnya, traf2 , diidentifikasi dalam PSR pada kromosom 17; namun, tidak ada SNP yang ditemukan berlokasi di wilayah gen untuk pengamatan perubahan frekuensi alel. traf2 terlibat dalam banyak jalur terkait imun, seperti jalur pensinyalan IL-17 dan jalur pensinyalan reseptor mirip NOD. Ia juga memainkan peran penting dalam proses apoptosis.
4 Diskusi
4.1 Efektivitas GS dalam Pemuliaan Selektif Ikan Croaker Kuning Besar terhadap C. irritans
Dengan menggunakan strategi sekuensing ddRAD, kami sebelumnya telah mengonfirmasi keefektifan GS dalam pemuliaan selektif ikan croaker kuning besar terhadap C. irritans (Zhao, Bai, dkk. 2021 ). Dalam penelitian saat ini, dua GS berturut-turut diterapkan untuk sifat resisten yang sama. Dibandingkan dengan penelitian GS sebelumnya, populasi dasar dan metode genotipe yang berbeda digunakan. Melalui uji tantangan, generasi F2 dan F3 menunjukkan kinerja resistensi yang lebih baik daripada kelompok kontrol. Praktik GS yang berulang sekali lagi telah membuktikan keunggulannya dalam pemuliaan galur ikan croaker kuning besar yang resisten terhadap C. irritans . Dibandingkan dengan ternak dan tanaman, penerapan GS pada hewan akuatik dimulai relatif terlambat, dan sebagian besar penelitian terbatas pada penggunaan validasi silang untuk menghitung akurasi GS, dengan demikian menunjukkan bahwa suatu sifat cocok untuk pemuliaan GS. Bukti paling langsung untuk membuktikan validitas GS adalah melalui pengujian keturunan kandidat seleksi. Saat ini ada dua cara umum untuk melakukan pengujian keturunan dari kandidat seleksi. Satu cocok untuk spesies di mana sejumlah besar keluarga dapat dibangun. Untuk sifat target tertentu, korelasi GEBV mid-parent dengan kinerja keturunan rata-rata untuk setiap keluarga pengujian keturunan digunakan untuk mengevaluasi prediktabilitas model GS. Roger L. Vallejo telah melakukan banyak karya penelitian representatif, seperti GS untuk resistensi penyakit air dingin bakteri pada ikan trout pelangi (Vallejo et al. 2016 , 2017 ) dan resistensi kutu laut pada strain akuakultur salmon Atlantik Amerika Utara (Vallejo et al. 2024 ). Karya-karya ini telah sangat memvalidasi efektivitas GS dalam pemuliaan selektif sifat-sifat tahan penyakit kompleks pada hewan air. Jenis lain dari pengujian keturunan kandidat seleksi lebih cocok untuk spesies di mana sejumlah besar keluarga tidak dapat dibangun, yang mengukur perolehan kinerja rata-rata keturunan dari kandidat seleksi dengan GEBV tinggi relatif terhadap keturunan populasi yang tidak dipilih. Mengingat ukuran ini, tim kami telah melakukan banyak studi penelitian perintis dan representatif tentang pemuliaan selektif sifat-sifat kompleks pada ikan kerapu kuning besar, seperti ketahanan terhadap bintik putih (Zhao, Bai, et al. 2021 ), ketahanan terhadap bintil putih visceral (Bai, Wang, Zhao, Ke, et al. 2022 ), kinerja renang yang unggul (Zeng et al. 2023 ), dan pemanfaatan protein nabati yang efisien (Ke et al. 2022 ). Sebagai kesimpulan, kami telah berulang kali menunjukkan efisiensi dan kepraktisan GS dalam pemuliaan selektif ikan kerapu kuning besar.
4.2 GS dengan Cepat Mendorong Divergensi Struktur Genetik Melalui Tiga Generasi Berturut-turut
Dalam studi saat ini, GEBV diperiksa untuk meningkat secara bertahap dengan generasi, dan keragaman genetik menunjukkan sedikit penurunan. Koefisien perkawinan sedarah ( F roh) meningkat secara bertahap dengan generasi. Dengan menggunakan set data sapi perah AS yang sangat besar yang berisi 154.008 banteng dan 33.022.242 sapi yang lahir sejak 1975, para peneliti telah memeriksa perolehan genetik (diukur sebagai nilai pembiakan yang diprediksi, PBV) dari sifat-sifat produksi, seperti susu, lemak, dan protein (Guinan et al. 2023 ). Hasil penelitian di atas menunjukkan bahwa PBV dari sifat-sifat produksi telah meningkat dengan mantap dari waktu ke waktu, khususnya pada ras Holstein dan Jersey, yang paling diuntungkan dari GS, dengan peningkatan perolehan genetik hingga 192% sejak GS diterapkan pada tahun 2009. Dampak negatifnya adalah perkawinan sedarah genomik meningkat pada banteng dan sapi Holstein, yang konsisten dengan hasil kami. Secara luas diterima bahwa seleksi buatan jangka panjang dapat menyebabkan hilangnya keragaman genetik. Namun, jika durasi seleksi pendek, banyak penelitian menunjukkan hasil yang tidak konsisten. Dalam program pembiakan selektif massal untuk tiram Kumamoto, generasi F4 menunjukkan variasi genetik yang secara signifikan lebih rendah dibandingkan dengan F0 (Ma et al. 2023 ). Penurunan keragaman genetik diamati dalam proses tiga seleksi massa berturut-turut untuk sifat pertumbuhan pada abalon Pasifik (N. Chen et al. 2017 ); namun, studi penelitian independen lainnya menemukan bahwa variasi genetik selama tiga generasi dipertahankan pada galur cangkang hitam abalon Pasifik, yang dikaitkan dengan penggunaan rasio jenis kelamin yang seimbang dan ukuran induk yang besar (Xu et al. 2019 ). Dibandingkan dengan populasi liar, keragaman genetik yang melimpah dari galur terpilih di Crassostrea nippona tidak menunjukkan kehilangan yang terdeteksi selama tiga generasi seleksi massa (Hu et al. 2022 ). Sejauh ini, belum ada laporan tentang perubahan keragaman genetik galur terpilih di bawah GS. Hasil penelitian kami menunjukkan bahwa keragaman nukleotida mengalami penurunan selama dua GS berturut-turut tetapi tidak mencapai tingkat yang signifikan. Di masa mendatang, seleksi berkelanjutan untuk ketahanan terhadap bercak putih harus diadopsi lebih banyak strategi untuk mengendalikan hilangnya keragaman genetik dan peningkatan koefisien perkawinan sedarah, seperti memperluas skala induk dan menghindari perkawinan antara kerabat dekat.
Telah diketahui secara umum bahwa domestikasi atau seleksi buatan jangka panjang untuk sifat target akan mengarah pada divergensi struktur genetik populasi. Sebagian besar penelitian difokuskan pada status akhir struktur populasi setelah seleksi jangka panjang, tetapi sedikit penelitian yang menyelidiki apakah struktur genetik berubah pada tahap awal seleksi atau bagaimana struktur genetik bergeser dari generasi ke generasi dengan menganalisis data genotipe historis. Dalam penelitian ini, analisis struktur genetik mengungkapkan bahwa diferensiasi genetik terjadi di antara tiga generasi berturut-turut. Hasil dari dua penelitian di atas tidak menemukan diferensiasi genetik yang signifikan dalam tiga generasi seleksi massa untuk sifat pertumbuhan pada abalon Pasifik (Xu et al. 2019 ) dan Crassostrea nippona (Hu et al. 2022 ). Tidak adanya diferensiasi genetik yang terdeteksi kemungkinan besar disebabkan oleh jumlah penanda genetik dan individu eksperimen yang sangat terbatas, yang mungkin tidak cukup kuat untuk mengevaluasi diferensiasi genetik. Sebaliknya, sejumlah besar penanda SNP dan individu eksperimen disertakan dalam penelitian ini, memastikan bahwa bahkan diferensiasi genetik yang halus dapat dideteksi. Yang terpenting, kami menemukan bahwa arah pergeseran struktur genetik dalam dua GS berturut-turut konsisten, dan arah perubahannya sesuai dengan yang kami harapkan, yaitu bergerak ke arah para penyintas generasi F1. Hasil kami menunjukkan bahwa pemanfaatan GS dapat menyebabkan pergeseran struktur genetik yang dapat dideteksi hanya dalam beberapa generasi.
4.3 Jalur Sinyal HIF-1 yang Terlibat dalam Seleksi Berturut-turut untuk Resistensi Bercak Putih pada Ikan Croaker Kuning Besar
Kami telah mengungkapkan bahwa diferensiasi genetik yang signifikan dan bertahap terjadi pada dua GS berturut-turut, yang menunjukkan bahwa seleksi buatan secara teoritis akan menghasilkan tanda-tanda seleksi genomik. Berdasarkan teori seleksi positif, banyak PSR diidentifikasi melalui integrasi berbagai informasi dan analisis komprehensif, dan jalur biologis serta gen yang terlibat dalam PSR kemungkinan besar akan dikaitkan dengan resistensi bercak putih. Jalur pensinyalan HIF-1 (faktor 1 yang dapat diinduksi hipoksia) diperkaya dalam PSR yang dipertahankan setelah penyaringan berdasarkan empat kriteria berbeda. Respons transkripsi utama terhadap stres hipoksia dimediasi oleh jalur pensinyalan HIF-1 (Majmundar et al. 2010 ). Theront C. irritans sering menjadi parasit pada jaringan insang inang, yang menyebabkan kerusakan pada integritas struktur filamen insang, sehingga memengaruhi asupan oksigen dan menyebabkan ketidakseimbangan dalam homeostasis oksigen. Oleh karena itu, jalur pensinyalan HIF-1 diinduksi untuk mempertahankan homeostasis oksigen dan mengatur jalur terkait metabolik hilir untuk menghasilkan lebih banyak energi yang diperlukan untuk fungsi dasar. Dalam GWAS kami sebelumnya dari ikan croaker kuning besar terhadap C. irritans , jalur pensinyalan HIF-1 juga dianotasi dalam daerah QTL yang terdeteksi (Zhao, Zhou, et al. 2021 ). Analisis transkriptom sebelumnya dari ikan croaker kuning besar sebagai respons terhadap infeksi C. irritans juga menemukan bahwa jalur pensinyalan HIF-1 diperkaya dalam insang (Bai, Wang, Zhao, Bai, et al. 2022 ). Secara keseluruhan, jalur pensinyalan HIF-1 dapat memainkan peran penting dalam resistensi C. irritans pada ikan croaker kuning besar. Sebagai gen inti dalam jalur pensinyalan HIF-1, famili HIF-1 tidak ditemukan dalam PSR. Namun, gen penting lainnya, crebbp , terdeteksi di PSR pada kromosom 1, yang telah dikonfirmasi sebagai komponen kunci yang terlibat dalam jalur pensinyalan HIF-1. Crebbp memiliki fungsi asetiltransferase intrinsik dan ada di mana-mana pada vertebrata. Domain pengikatannya yang kaya memungkinkannya untuk berpartisipasi dalam banyak jaringan regulasi dan mengatur ekspresi ribuan gen, dengan demikian mempertahankan homeostasis sel dan membantu mengatasi perubahan di lingkungan eksternal (Sheikh dan Akhtar 2019 ). Dalam kondisi hipoksia, protein Hif-1α yang terakumulasi pertama-tama akan merekrut Crebbp melalui domain C-TAD-nya, kemudian membentuk dimer dengan Hif-1β melalui domain bHLH untuk menjadi kompleks transkripsi fungsional. Akhirnya, ia mengikat elemen respons hipoksia (HRE) pada gen target untuk akhirnya memulai ekspresi (Zimna dan Kurpisz 2015 ). Penanda SNP ditemukan jatuh ke dalam intron crebbp, dan frekuensi alel SNP ini meningkat pesat pada setiap generasi, yang menunjukkan bahwa crebbp mengalami seleksi kuat. Secara kolektif, crebbp merupakan gen kandidat penting yang terkait dengan resistensi terhadap C. irritans , dan validasi fungsional molekuler lebih lanjut diperlukan di masa mendatang.
4.4 Jalur Biologi Penting Lainnya yang Terlibat dalam Seleksi Berturut-turut untuk Ketahanan terhadap Bercak Putih pada Ikan Croaker Kuning Besar
Sementara jalur pensinyalan HIF-1 menerima perhatian khusus karena perannya yang mapan dalam respons hipoksia selama parasitisme insang, beberapa jalur penting lainnya perlu didiskusikan karena kemungkinan berkontribusi pada fenotipe resistensi yang kompleks. Jalur metabolisme energi yang diidentifikasi dalam PSR, termasuk jalur pensinyalan Apelin, Fosfolipase D, dan Glukagon, menunjukkan bahwa modulasi homeostasis energi sangat penting untuk kelangsungan hidup inang selama infeksi. Jalur pensinyalan Apelin mengatur fungsi kardiovaskular dan metabolisme energi (Chng et al. 2013 ), dan seleksinya dapat mencerminkan adaptasi terhadap stres kardiovaskular yang disebabkan oleh kerusakan insang. Demikian pula, jalur pensinyalan Glukagon, yang mengatur metabolisme glukosa selama kondisi puasa atau stres (Jiang dan Zhang 2003 ), dapat membantu mempertahankan pasokan energi selama peningkatan tuntutan metabolik dari aktivasi respons imun. Seleksi dalam jalur ini menunjukkan bahwa pemanfaatan dan distribusi energi yang efisien mungkin sama pentingnya untuk kelangsungan hidup seperti respons imun langsung. Mengenai jalur terkait imun, identifikasi jalur pensinyalan reseptor mirip NOD di PSR sangat penting. Reseptor mirip NOD berfungsi sebagai sensor intraseluler yang mendeteksi pola molekuler terkait patogen dan pola molekuler terkait kerusakan, yang memulai respons inflamasi melalui aktivasi pensinyalan NF-κB dan MAPK (Motta et al. 2015 ). Tekanan seleksi pada jalur ini menunjukkan bahwa pengenalan patogen yang ditingkatkan dan regulasi inflamasi yang tepat mungkin merupakan komponen utama resistensi terhadap C. irritans . Selain itu, keberadaan jalur pensinyalan reseptor sel B di PSR menunjukkan bahwa pengembangan imunitas adaptif mungkin berperan dalam resistensi, berpotensi melalui peningkatan produksi antibodi terhadap parasit (Xu et al. 2016 ). Pemilihan tanda tangan dalam jalur kematian sel, seperti Autophagy dan Apoptosis, menyoroti pentingnya kematian sel yang terkendali dalam respons inang. Selama infeksi C. irritans , jalur-jalur ini kemungkinan memiliki dua tujuan: eliminasi sel yang terinfeksi atau rusak dan pengaturan peradangan (Mariño et al. 2014 ). Keseimbangan antara apoptosis dan autofagi pada jaringan yang terinfeksi dapat menentukan apakah inang dapat menahan infeksi sambil mempertahankan fungsi jaringan yang memadai.
Di luar jalur spesifik, identifikasi kami terhadap TRAF2 di bawah tekanan seleksi yang kuat memberikan wawasan ke dalam mekanisme molekuler resistensi. TRAF2 adalah protein adaptor penting dalam pensinyalan reseptor TNF yang mengatur aktivasi NF-κB, pensinyalan JNK, dan keputusan kelangsungan hidup sel (Wajant dan Scheurich 2001 ). Seleksinya menunjukkan bahwa penyempurnaan respons inflamasi dan regulasi kematian sel mungkin berada di bawah tekanan evolusi selama seleksi buatan untuk resistensi. Studi terbaru pada ikan telah menunjukkan bahwa TRAF2 memodulasi respons antivirus dan peradangan (Eslamloo et al. 2017 ), dan seleksinya dalam studi kami menunjukkan kepentingan serupa dalam pertahanan antiparasit. Identifikasi jalur beragam ini di bawah seleksi menunjukkan bahwa resistensi terhadap infeksi C. irritans melibatkan interaksi kompleks dari metabolisme energi, aktivasi imun, dan regulasi kematian sel. Arsitektur genetik multifaset ini selaras dengan studi transkriptomik sebelumnya yang menunjukkan respons fisiologis luas terhadap infeksi (Zhang et al. 2018 ) dan menjelaskan mengapa pemuliaan untuk resistensi merupakan tantangan tetapi dapat dicapai melalui pendekatan seleksi genom yang menangkap interaksi genetik yang kompleks ini.
5. Kesimpulan
Dalam upaya membudidayakan strain yang resistan untuk mengurangi kerugian ekonomi yang disebabkan oleh penyakit bercak putih yang parah, kami menggunakan dua GS berturut-turut untuk menghasilkan tiga generasi berturut-turut. Tingkat kelangsungan hidup yang meningkat secara signifikan yang diamati pada generasi F2 dan F3 setelah uji tantangan C. irritans sangat mendukung efektivitas dan penerapan GS dalam meningkatkan sifat resistensi. Analisis struktur genetik menunjukkan bahwa hanya dua generasi GS berturut-turut yang cukup untuk menginduksi divergensi genetik yang substansial dalam populasi. Lebih jauh, pemindaian genom secara luas untuk tanda-tanda seleksi mengidentifikasi beberapa wilayah kandidat yang berpotensi mengalami seleksi buatan. Di antara ini, crebbp, gen yang terlibat dalam jalur pensinyalan HIF-1, muncul sebagai kandidat utama yang terkait dengan resistensi terhadap C. irritans . Selain jalur HIF, kami juga menyoroti jalur biologis lain yang diperkaya terkait dengan respons imun, metabolisme energi, dan kematian sel, yang mencerminkan arsitektur poligenik kompleks yang mendasari resistensi. Temuan-temuan ini secara kolektif memberikan wawasan baru ke dalam mekanisme genetik yang mendorong resistensi penyakit dan menunjukkan kekuatan GS sebagai alat praktis untuk meningkatkan resistensi penyakit pada spesies akuakultur.



