Abstrak
Mengidentifikasi daerah genomik dalam seleksi sangat penting untuk memahami sejarah evolusi ayam domestik. Ayam asli Semenanjung Arab (AP) sebagian besar ditemukan di luar ruangan, dipelihara bersama ternak lain untuk tujuan produksi. Burung-burung ini menunjukkan ketahanan tinggi terhadap suhu ekstrem (panas dan dingin), khas lingkungan gurun. Tekanan seleksi yang bertanggung jawab atas adaptasi lokal yang unik pada burung-burung ini sebagian besar masih belum teridentifikasi. Di sini, kami bertujuan untuk menyelidiki keragaman genom dan struktur 15 populasi ayam asli termasuk 13 populasi dari AP ( n = 5), Ethiopia ( n = 6), dan Republik Rakyat Tiongkok ( n = 2). Kami juga menyertakan dua populasi ayam komersial, Fayoumi (dipilih untuk toleransi panas) dan Chantecler (dikenal karena toleransi dinginnya). Analisis komponen utama (PC) memisahkan semua populasi berdasarkan daerah asal geografisnya. PC1 memisahkan populasi Ethiopia dari populasi Tiongkok dan AP, sementara PC2 memisahkan populasi AP dari Chantecler, dan populasi Ethiopia dari Dulong dan Chantecler. Tanda-tanda genom secara luas dari analisis mengidentifikasi banyak kandidat daerah di bawah seleksi positif. Mereka termasuk gen yang mungkin terkait dengan termotoleransi. Ini terlibat dalam keseimbangan energi dan metabolisme ( SUGCT , HECW1 , MMADHC ), apoptosis sel ( APP , SRBD1 , NTN1 , PUF60 , SLC26A8 , DAP , SUGCT ), angiogenesis ( RYR2 , LDB2 , SOX5 ), perlindungan kulit terhadap radiasi matahari ( FZD10 , BCO2 , WNT5B , COL6A2 , SIRT1 ) serta pertumbuhan ( NELL1 ). Temuan kami menunjukkan bahwa populasi ayam Arab memiliki polimorfisme kumpulan gen yang berbeda dalam kaitannya dengan adaptasi mereka terhadap lingkungan iklim yang keras di AP.
PERKENALAN
Populasi ayam asli, yang terbentuk selama ribuan tahun melalui adaptasi, menawarkan wawasan menarik tentang tanda-tanda genomik seleksi alam dalam menanggapi lingkungan termal yang bervariasi. Burung-burung yang mudah beradaptasi ini telah mengembangkan sifat-sifat unik yang memungkinkan mereka untuk bertahan hidup dan bereproduksi dalam iklim yang beragam, menyoroti interaksi rumit antara evolusi genetik dan tekanan lingkungan (Lawler, 2016 ). Misalnya, Perini et al. ( 2020 ) meninjau jalur molekuler yang terkait dengan stres panas, sementara Juiputta et al. ( 2023 ) membahas strategi genetik untuk meningkatkan toleransi panas dan mempertahankan produktivitas dalam peternakan unggas tropis. Selain itu, Nawaz et al. ( 2024 ) menilai gen dan jalur penting yang terkait dengan toleransi panas, seperti respons syok panas, sistem pertahanan antioksidan, fungsi imun, dan homeostasis seluler.
Mereka tumbuh subur di iklim panas, seperti daerah kering di sub-Sahara Afrika atau gurun terik di Jazirah Arab (AP), setelah mengembangkan serangkaian adaptasi untuk menahan tekanan panas yang tiada henti (Fathi, Al-Homidan, Abou-Emera & Al-Moshawah, 2017 ; Kanyama et al., 2022 ). Adaptasi ini mencakup berbagai polimorfisme fisiologis, perilaku, dan genetik yang ditujukan untuk mempertahankan homeostasis di lingkungan bersuhu tinggi (Cheng & Muir, 2005 ). Yang menonjol di antaranya adalah mekanisme termoregulasi yang efisien, seperti peningkatan reflektansi bulu dan kapasitas untuk menghilangkan panas melalui perilaku seperti mencari tempat teduh dan terengah-engah (Soleimani et al., 2011 ).
Studi genomik telah mengungkap tanda tangan genetik yang terkait dengan toleransi panas, menekankan variasi substansial dalam gen yang bertanggung jawab untuk beradaptasi dengan stres panas di antara populasi ayam asli. Misalnya, Guo et al. ( 2022 ) mengidentifikasi beberapa sapuan selektif yang mengandung gen dalam seleksi ( FABP2 , RAMP3 , SUGCT , dan TSHR ) pada ayam asli dari daerah tropis (Tiongkok utara, Indonesia, Sri Lanka, dan Thailand), yang mungkin terkait dengan adaptasi terhadap suhu sekitar yang lebih tinggi. Mereka secara khusus menemukan mutasi salah arti pada TSHR yang dapat meningkatkan toleransi panas pada ayam. Zhuang et al. ( 2020 ) mengidentifikasi gen yang terkait dengan termotolerans pada ayam asli Taiwan, termasuk gen yang terlibat dalam perbaikan DNA, respons stres seluler, apoptosis, dan stres oksidatif metabolik. Lawal et al. ( 2018 ) mengamati bahwa gen KCNMA1 berada dalam wilayah tanda tangan seleksi pada populasi ayam Arab Saudi dan Sri Lanka. Gen ini terkait dengan respons terhadap tantangan hipoksia dan regulasi kontraksi otot polos. Beberapa gen yang terlibat dalam adaptasi terhadap iklim juga diidentifikasi pada ayam kampung Tiongkok, termasuk APP , FABP1 , SMYD1 , UBE4B , NALCN , PDGFRA , NRP1 , CORIN , CLPTM 1L , CRADD , PARK2 , SIM2 , AHR , ESRRG , IL18 , dan BVES (Gu et al., 2020 ). Pada ayam kampung Nigeria, gen yang mungkin terkait dengan adaptasi terhadap stres panas (misalnya, ILF3 , HSF1 , SLC44A2 , SFTPB , HIF3A , CDC37 , dan TSHR ) baru-baru ini dilaporkan (Rachman et al., 2024 ).
Selain itu, investigasi genomik telah mengidentifikasi varian genetik yang terkait dengan adaptasi dingin, termasuk varian yang mengatur perkembangan bulu, metabolisme lipid, dan termogenesis yang disebabkan oleh dingin (Wang et al., 2015 ). Adaptasi ini sangat diperlukan untuk bertahan hidup di iklim dingin, di mana konservasi energi dan retensi panas sangat penting. Dalam analisis genom yang komprehensif oleh Xu et al. ( 2021 ), dua kandidat daerah terpilih mencakup gen yang mungkin terkait dengan adaptasi terhadap stres dingin di Chantecler telah diidentifikasi. Daerah ini meliputi gen ME3 , yang terlibat dalam metabolisme lemak, dan ZNF536 , yang terkait dengan sistem saraf.
Keadaan iklim AP menawarkan peluang luar biasa untuk mengungkap determinan genetik yang mendorong adaptasi terhadap kondisi lingkungan yang menantang. AP menunjukkan beragam pola iklim musiman dan regional. Secara khusus, wilayah tertentu mengalami suhu musim panas yang terik yang melebihi 50°C (122°F), yang mengakibatkan lingkungan yang sangat panas dan kering (Abou-Shaara et al., 2013 ). Sebaliknya, suhu dapat anjlok hingga beku selama musim dingin, terutama di wilayah utara. Hebatnya, meskipun menghadapi tantangan lingkungan yang berat, ayam lokal tidak hanya bertahan hidup tetapi juga hidup berdampingan secara harmonis dengan manusia dalam kondisi yang keras ini (Fathi, Al-Homidan, Motawei, et al., 2017 ; Soliman et al., 2016 ). Tanpa sumber statistik yang komprehensif tentang ukuran populasi mereka atau jumlah peternakan yang membesarkan mereka, nasib sumber daya genetik ayam lokal di AP masih belum pasti. Fathi, Al-Homidan, Motawei, et al. ( 2017 ) menyoroti bahwa ras asli berada pada risiko kepunahan akibat erosi genetik yang disebabkan oleh perkawinan silang yang tidak terkendali dengan ras eksotik, yang semakin diperburuk oleh praktik manajemen yang tidak memadai.
Di sini, kami menyelidiki ayam lokal dari AP yang bertujuan untuk menjembatani kesenjangan pengetahuan dalam keragaman genom ayam dan adaptasi di wilayah tersebut. Meskipun ada bukti observasional bahwa populasi ayam lokal beradaptasi dengan baik terhadap lingkungan yang menantang, mekanisme genetik yang mendorong adaptasi ini masih kurang dipelajari (Muchadeyi & Dzomba, 2017 ). Selain itu, upaya minimal telah dilakukan untuk membedah keragaman genetik dan susunan ayam lokal di wilayah AP.
BAHAN DAN METODE
Pengambilan sampel dan pengurutan
Urutan genom dari 156 sampel dari 15 populasi ayam asli dari berbagai wilayah geografis yang mencakup zona iklim yang berbeda (Tabel 1 dan Tabel S1 ) disertakan dalam penelitian ini. Sepuluh dari populasi ini telah dipelajari sebelumnya, sementara lima populasi adalah data baru. Ini termasuk Fayoumi ( n = 10), yang berasal dari Mesir, Black feather (BL-KFU; n = 13), dan Brown feather (BR-KFU; n = 15), yang dibesarkan di King Faisal University Research and Training Station di wilayah timur Kerajaan Arab Saudi. Burung Fayoumi mencakup delapan betina dan dua jantan, yang dipilih secara acak dari kawanan yang berjumlah 700 ekor burung. Burung BL-KFU dan burung BR-KFU masing-masing mencakup 10 betina dan tiga jantan, yang dipilih secara acak dari kawanan yang berjumlah 500 ekor burung dan 1000 ekor burung. Selain itu, kami mengambil sampel burung asli di tingkat peternakan untuk dua populasi asli. Burung-burung ini adalah Buqyiq (BU-VI; n = 13) dari kota Buqyiq di Arab Saudi dan burung Oman ( n = 20) dari Negara Bagian Barka di Oman. BU-VI mencakup 10 betina dan tiga jantan. Mereka diambil sampelnya dari satu peternakan dengan populasi ayam lokal sebanyak 100 ekor. Semua ayam Oman adalah betina. Data genom dari sampel yang tersisa diunduh dari basis data genom publik (Tabel S1 ). Koordinat sistem penentuan posisi geografis dicatat untuk populasi baru (lihat Tabel S1 ). Untuk genom yang baru diurutkan, sampel darah dikumpulkan dari vena sayap dalam tabung EDTA. DNA diekstraksi menggunakan Qiagene DNeasy Blood and Tissue Kits mengikuti protokol pabrik pembuatnya ( https://www.qiagen.com/ca/resources/download.aspx?id=63e22fd7-6eed-4bcb-8097-7ec77bcd4de6&lang=en ) dan disuspensikan kembali pada konsentrasi akhir 50 ng/μl. DNA diurutkan di NEOGEN ( https://www.neogen.com ) di Inggris untuk pengurutan genom lengkap pada cakupan 20x pada platform HiSeq Illumina.
| Populasi | Ukuran sampel | Kisaran suhu di lokasi pengambilan sampel tahun 2020–2021 https://www.worldclim.org/ | Ketinggian (m) | Rata-rata curah hujan (mm) | Klasifikasi ekologi ( https://weatherandclimate.com/countries ) | Wilayah geografis (negara) | Referensi | Nomor akses proyek ENA | |
|---|---|---|---|---|---|---|---|---|---|
| Minimum (°C) | Maksimum (°C) | ||||||||
| Populasi iklim panas | |||||||||
| Bahasa Oman | 20 | 19 | 42 | tahun 1200 | 80–100 | Gurun subtropis/kering | Oman—Negara Barka | Urutan baru | PRJEB77573 |
| orang Arab Saudi | 5 | 17 | 43 | 154 | 74 | Gurun subtropis/kering | Arab Saudi | Lawal dkk. ( 2018 ) | PRJNA453469 |
| Fayoumi | 10 | Sampel populasi Mesir di Alhufuf (Arab Saudi) | Urutan baru | PRJEB77573 | |||||
| BR-KFU | 15 | Alhufuf (Arab Saudi) | |||||||
| BL-KFU | 13 | ||||||||
| BU-VI | 12 | 90 | |||||||
| Populasi iklim dingin | |||||||||
| Pelantun | 9 | -25 | 20 | tahun 1652 | >1000 | Dingin/sedikit lembab | Quebec (Kanada) | Xu dan kawan-kawan ( 2021 ) | PRJNA720223 |
| Dulong | 10 | -6 | 16 | 3000 | >3000 | Dinginnya dataran tinggi tropis | Sabuk Ngarai Alpen di Pegunungan Hengduan Utara–Selatan (Republik Rakyat Tiongkok) | Wang dkk. ( 2020 ) | PRJNA559932 |
| Tibet | 5 | -7 | 23 | 3650 | – | Dingin/kering | Dataran Tinggi Tibet (Republik Rakyat Tiongkok) | Zhang dkk. ( 2016 ) | PRJNA309581 |
| Populasi Ethiopia di daerah dingin | |||||||||
| Alfa Midir | 9 | 1 | 19 | 3451 | >1000 | Tropis dingin/sub-lembab | Pegunungan Amhara Tengah (Ethiopia) | Gheyas dan kawan-kawan ( 2022 ) | PRJEB39275 |
| Gafera | 10 | 5 | 25 | tahun 2592 | 900–1000 | ||||
| Negasi Amba | 10 | 3 | 20 | 3071 | 900–1000 | ||||
| Populasi Ethiopia di daerah hangat | |||||||||
| Gesses | 10 | 11 | 33 | tahun 1546 | >900 | Daerah tropis hangat/semi-kering | Lembah Amhara Barat (Etiopia) | Gheyas dan kawan-kawan ( 2022 ) | PRJEB39275 |
| Pelukan | 9 | 15 | 37 | 979 | >900 | Afar Timur (Ethiopia) | |||
| Anak kecil | 9 | 11 | 33 | tahun 1418 | >900 | Daerah tropis hangat/kering | Lembah Amhara Barat (Etiopia) | ||
Pemetaan dan pemanggilan varian
Pembacaan sekuens genom secara keseluruhan dipetakan ke rakitan genom referensi “GRCg6a”, menggunakan algoritma bwa-mem dari burrows-wheeler aligner versi 0.7.17 (Li & Durbin, 2009 ). Pembacaan yang dipetakan diurutkan berdasarkan koordinat dan duplikat PCR ditandai dan dihapus menggunakan opsi SortSam dan MarkDuplicates dari picard versi 2.18.29 ( https://broadinstitute.github.io/picard/ ), masing-masing. Setiap sampel kemudian dikenakan kalibrasi ulang skor kualitas dasar menggunakan “situs yang diketahui” dbSNP ( http://ftp.ensembl.org/pub/release-104/variation/vcf/gallus_gallus/ ). Proses ini ditujukan untuk menghilangkan kesalahan atau ketidakselarasan selama pengurutan dan mengidentifikasi varian yang diketahui. Polimorfisme nukleotida tunggal (SNP) dipanggil menggunakan algoritma HaplotypeCaller dari genome analysis toolkit (gatk ) versi 4.2.2.0 pada mode GVCF (McKenna et al., 2010 ) pada setiap sampel. Tempat kerja untuk setiap kromosom diterapkan menggunakan algoritma GenomicsDBImport . Genotipe gabungan dilakukan untuk mengidentifikasi varian pada semua sampel secara bersamaan diikuti oleh penyaringan keras menggunakan algoritma VariantFiltration untuk pemanggilan SNP berkualitas tinggi. Selanjutnya, varian autosomal bi-allelic yang lolos langkah penyaringan varian dipilih untuk analisis hilir.
Keanekaragaman genom dan struktur populasi
Beberapa metode digunakan untuk mengeksplorasi keragaman genetik dan struktur populasi. vcftools versi 0.1.14 (Danecek et al., 2011 ) digunakan untuk estimasi keragaman genetik pada tingkat genom, termasuk diferensiasi populasi berpasangan ( F ST ) dan heterozigositas. Rata-rata genom F ST dihitung menggunakan jendela 20-kb dengan langkah geser 10 kb. Analisis kontrol kualitas dilakukan pada SNP autosomal yang disebut untuk tujuan analisis keragaman genetik dan struktur populasi menggunakan plink 1.9 (Chang et al., 2015 ). SNP dengan frekuensi alel minor ≤ 0,05 (4.289.890 SNP), dan tingkat panggilan genotipe ≤ 95% dikecualikan (4.143 SNP). Untuk analisis keragaman genetik, SNP berpasangan dengan ketidakseimbangan hubungan tinggi (koefisien korelasi kuadrat r 2 > 0,1) selanjutnya dikecualikan (17.237.390 SNP). Analisis komponen utama dilakukan dengan 3.374.709 SNP yang tersisa menggunakan opsi –pca dalam plink 1.9 dan diplot menggunakan paket r tidyverse (Wickham et al., 2019 ). Program admixture versi 1.3.0 (Alexander et al., 2009 ), untuk hingga 12 klaster leluhur yang disimpulkan ( K ), digunakan untuk menilai proporsi leluhur genom yang optimal dalam populasi ayam yang dianalisis.
Tanda-tanda analisis seleksi
Kami melakukan empat tes di semua populasi untuk mendeteksi tanda-tanda kandidat seleksi positif termasuk heterozigositas gabungan Hp (Rubin et al., 2010 ), skor haplotipe terintegrasi iHS (Voight et al., 2006 ), diferensiasi populasi F ST (Akey et al., 2002 ), dan homozigositas haplotipe lintas populasi yang diperluas XP-EHH (Sabeti et al., 2007 ). Kami menggunakan jendela geser 20-kb dengan langkah 10-kb di semua metode dengan minimal 20 SNP per jendela. Nilai Hp dan F ST distandarisasi ( ZHp dan ZF ST ) untuk memastikan konsistensi di semua analisis. Analisis iHS dan XP-EHH dilakukan menggunakan perangkat lunak HAPBIN (Ahbara et al., 2022 ; Maclean et al., 2015 ) setelah menghilangkan SNP dengan genotipe yang hilang. Analisis iHS dan XP-EHH dilakukan untuk SNP individual, dan kemudian nilai rata-rata dihitung dalam jendela untuk metrik iHS standar ( iHS _std) dan XP-EHH ( XP-EHH _std).
Identifikasi gen dan anotasi fungsional
Kami menggunakan rakitan genom referensi “GRCg6a”, rilis 110 basis data dbSNP, (2023), dan “VEP” Ensembl (McLaren et al., 2016 ) untuk posisi SNP dan identifikasi gen. Ambang batas untuk deteksi outlier melibatkan pemilihan persentil terendah ekstrem 0,001 dari ZHp , dan persentil tertinggi ekstrem 0,001 untuk nilai iHS , ZF ST , dan XP-EHH , dari distribusi empiris masing-masing.
Wilayah yang diidentifikasi kemudian dikonsolidasikan menggunakan bedtools v.2.25.0. Gen yang tumpang tindih dengan wilayah kandidat diidentifikasi berdasarkan basis data 108 Ensembl Genes menggunakan alat Ensembl BioMart daring ( http://www.ensembl.org/biomart ). Dua daftar terpisah dibuat: (i) gen yang tumpang tindih atau dalam tanda tangan wilayah seleksi menggunakan analisis ZHp dan iHS dalam populasi ; dan (ii) gen yang tumpang tindih atau dalam tanda tangan seleksi yang diidentifikasi oleh perbandingan berpasangan F ST dan XP-EHH antar populasi . Kami menggunakan perangkat lunak Database for Annotation, Visualization, and Integrated Discovery (DAVID) versi 6.8 ( https://david.ncifcrf.gov/summary.jsp ) untuk meneliti daftar gen untuk pengayaan gen yang signifikan yang terkait dengan kategori fungsional tertentu. Analisis DAVID mencakup berbagai kategori anotasi, seperti Gene Ontology (GO), Proses Biologis, dan Fungsi Molekuler GO, untuk mengidentifikasi proses dan fungsi biologis yang diperkaya dalam daftar gen. Ambang signifikansi nilai p yang dikoreksi Benjamini sebesar 0,05 digunakan untuk menentukan pengayaan statistik. Lebih jauh, kami menyelidiki peran biologis setiap gen yang dianotasi melalui penelusuran literatur ekstensif yang menggabungkan informasi dari berbagai spesies.
HASIL
Pengurutan dan analisis varian
Urutan genom sampel ayam dari Arab Saudi, Oman, dan Mesir menghasilkan rata-rata 221–480 juta pembacaan urutan berpasangan. Cakupan genom rata-rata untuk urutan tersebut berkisar antara 16,98× hingga 57,46× (lihat Tabel S2 ). Pembacaan tersebut diselaraskan dengan genom referensi ayam (GRCg6a), dan analisis gabungan dari semua sampel kemudian dilakukan. Analisis ini mengungkap sekitar 25 juta SNP berkualitas tinggi untuk 15 populasi. Menggabungkan semua populasi menemukan bahwa 38,25% dari seluruh set SNP (9,5 juta dari 24,9 juta) adalah baru menurut dbSNP rilis 110 (2023). Di antara populasi yang diperiksa, Chantecler memiliki persentase terendah (12,4%) SNP baru, dan populasi AP BL-KFU, BR-KFU, dan BU.VI memiliki yang tertinggi masing-masing 22,31%, 24,44%, dan 24,73% (Tabel S3 ). Kepadatan SNP yang diamati di seluruh kromosom diilustrasikan dalam Tabel S4 . Kepadatan rata-rata SNP di seluruh autosom adalah 26,49 SNP/kb. Kromosom 22 menunjukkan kepadatan SNP terendah, sedangkan kromosom 31 menunjukkan kepadatan tertinggi, diikuti oleh kromosom 33.
Struktur dan diferensiasi populasi
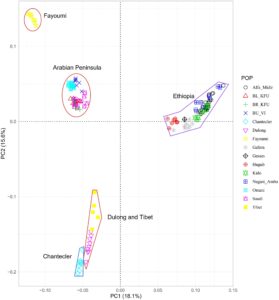
Struktur populasi diperiksa menggunakan analisis komponen utama, campuran dan analisis diferensiasi genetik ( FST ) . Komponen utama (PC)1, yang mencakup 18,1% variasi, secara jelas membedakan semua populasi Ethiopia dari populasi yang tersisa, sementara PC2, yang menjelaskan 15,6% dari total variasi, memisahkan populasi AP dan Fayoumi dari Chantecler, serta sebagian besar populasi Ethiopia dari Dulong dan Chantecler (Gambar 1 ). Populasi Oman dan Arab Saudi terletak pada posisi perantara pada Gambar 1. Analisis campuran ( K = 5) mendukung satu kumpulan gen leluhur untuk populasi Arab Saudi, sedangkan populasi Oman tampak jauh lebih bercampur (Gambar 2 ). Populasi lain menunjukkan susunan genetik yang berbeda, seperti yang ditunjukkan oleh latar belakang genetik mereka yang berbeda. Peta panas analisis F ST berpasangan (Tabel 2 ) menggambarkan bahwa diferensiasi populasi terendah diamati antara populasi Ethiopia Alfa Midir dan Negasi Amba ( F ST = 0,009), Gafera dan Gesses ( F ST = 0,022), Gesses dan Kido ( F ST = 0,022), dan Gafera dan Kido ( F ST = 0,024), serta antara populasi Cina Dulong dan Tibet ( F ST = 0,035). Sebaliknya, diferensiasi populasi tertinggi ditemukan antara Chantecler dan Fayoumi ( F ST = 0,349). Secara umum, ada diferensiasi genetik sedang antara populasi kecuali untuk Fayoumi dan Chantecler, yang menunjukkan nilai F ST tertinggi dengan populasi lainnya.

TABEL 2. Fiksasi populasi berpasangan ( FST ) di antara populasi ayam.

Mendeteksi sinyal sapuan selektif di seluruh genom
Empat pendekatan berbeda digunakan dalam uji seleksi untuk mengidentifikasi adaptasi termotoleransi (stres panas dan dingin). Pertama-tama kami bertujuan untuk mengidentifikasi kandidat daerah genom yang berpotensi di bawah seleksi positif untuk termotoleransi dalam setiap populasi secara terpisah. Daerah genom spesifik populasi dibandingkan di seluruh kelompok untuk mengevaluasi tumpang tindih antara populasi iklim panas (AP dan Fayoumi) dan populasi iklim dingin (Chantecler, Dulong, dan Tibet). Tujuannya adalah untuk mengidentifikasi kandidat daerah yang menunjukkan diferensiasi signifikan dalam frekuensi alel (menggunakan metode ZF ST ) atau dalam pola ketidakseimbangan hubungan (menggunakan metode XP-EHH ). Jendela kandidat diidentifikasi melalui analisis tanda tangan seleksi dengan persentil ekstrem 0,001, di mana ambang batas ditetapkan pada F ST standar ( ZF ST ) >3,24–6,24, dan XP-EHH standar absolut (| XP-EHH _std|) >3,24–4,11.
Wilayah kandidat dan gen yang terkait dengan adaptasi terhadap kondisi ekstrem dalam populasi
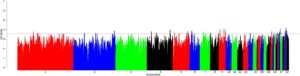
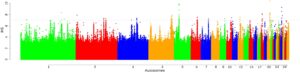
Kami menganalisis 81.007–95.110 dan 95.101–95.122 jendela di seluruh populasi dalam analisis ZHp dan iHS , masing-masing (Tabel S5 ). Sapuan ZHp yang signifikan terdeteksi di seluruh autosom genom ayam, kecuali pada kromosom 16, 19, 20, 21, 22, 25, 28, dan 31 (Gambar 3 dan Tabel S6 ). Sapuan iHS diamati di seluruh autosom kecuali kromosom 25, 27, dan 31 (Gambar 4 , Tabel S7 ). Di seluruh populasi AP di bawah tekanan panas (Oman, Arab Saudi, dan Fayoumi), jendela ini secara kolektif mencakup 169 gen ( ZHp ) dan 172 gen ( iHS ) Tabel 3 . Hasil terperinci disediakan dalam Tabel S8a–n untuk ZHp dan S9a–n untuk iHS , serta dalam Gambar S1a–l dan S2a–n . Gen kandidat menunjukkan fungsi biologis yang berbeda, khususnya terkait dengan toleransi panas, respons stres, imunitas, dan sistem saraf (Tabel S10 ). Analisis istilah GO dan jalur Kyoto Encyclopedia of Genes and Genomes (KEGG) yang terkait dengan gen-gen ini memberikan wawasan tambahan ke dalam potensi pentingnya mereka untuk jalur biologis yang berbeda, termasuk toleransi panas, pensinyalan adrenergik dalam kardiomiosit, respons terhadap bakteri, dan fosforilasi protein (Tabel S11 dan S12 ). Misalnya, RYR2 terlibat dalam takikardia ventrikel polimorfik yang diinduksi stres, dan yang menarik, itu terdeteksi pada populasi Tiongkok dalam kondisi dingin (Wang et al., 2015 ). LDB2 berkontribusi pada pembentukan pembuluh darah (Javerzat et al., 2009 ). Selain itu, APP , NTN3 , dan PUF60 dikaitkan dengan respons apoptotik terhadap stres (Gu et al., 2020 ), sementara CNTNAP2 berperan dalam proses sistem saraf (von Holdt et al., 2023 ) terdeteksi dalam populasi BL-KFU. Dalam populasi AP, ALX4 dan PLA2G15 , yang berperan dalam deposisi lemak dan proses metabolisme asam lemak (McManus et al., 2022 ; Zhang et al., 2016 ), terdeteksi bersama dengan COL6A2 , gen kunci dalam sintesis kolagen (Fleming et al., 2017 ; Wang et al., 2016 ). Menariknya, COL6A2Gen tersebut juga terdeteksi di wilayah genom yang menunjukkan sinyal ZHp negatif yang substansial (−4,67) di Gesses dan populasi Ethiopia lainnya, yang tinggal di lokasi tropis hangat/semi-kering. Lebih jauh, ORAOV1 dikaitkan dengan resistensi terhadap stres oksidatif, FGF19 membantu penyesuaian metabolisme selama puasa, dan PLCE1 berpotensi memengaruhi kandungan lemak intramuskular yang lebih tinggi.

| Bahasa Inggris CHR | Mulai (bp) | Akhir (bp) | Metode | Skor tertinggi | Gen dan Fungsinya |
|---|---|---|---|---|---|
| 5 | Nomor telepon 17 070 001 | 17.140.000 | Bahasa Indonesia: IHS | 8.24 | ORAOV1 terlibat dalam resistensi terhadap stres oksidatif (Togashi et al., 2014 ) |
| FGF19 memfasilitasi penyesuaian metabolisme selama puasa (Degirolamo et al., 2016 ) | |||||
| 6 | 20 920 001 | 20.940.000 | Bahasa Indonesia: IHS | 8.42 | PLCE1 terlibat dalam metabolisme (Li et al., 2022 ) |
| 1 | Nomor telepon 102 900 001 | Nomor telepon 102 920 001 | ZHp | -4,28 | APP mengatur apoptosis sel (Gu et al., 2020 ) |
| 2 | Nomor telepon 149 040 001 | Nomor telepon 149 060 001 | ZHp | -3,90 | PUF60 dikaitkan dengan apoptosis (Dong et al., 2019 ) |
| 5 | 21 340 001 | 21.390.000 | ZHp | -3,98 | ALX4 dikaitkan dengan penumpukan lemak (McManus et al., 2022 ) |
| 11 | 50 001 | 70.000 orang | ZHp | -3,76 | PLA2G15 dikaitkan dengan proses metabolisme asam lemak (Zhang et al., 2016 ) |
| 3 | Nomor telepon 37 070 001 | Nomor telepon 37 090 001 | ZHp | -3,94 | RYR2 memiliki peran dalam takikardia ventrikel polimorfik yang disebabkan oleh stres (Wang et al., 2015 ) |
| 2 | 53 320 001 | 53.340.000 | ZHp | -3,96 | Peran CNTNAP2 adalah dalam proses sistem saraf dan mungkin memainkan peran penting dalam bagaimana individu menyesuaikan diri dengan lingkungan baru (von Holdt et al., 2023 ) |
Tiga belas dari 133 gen yang terdeteksi di ZHp dan iHS mungkin terkait dengan adaptasi terhadap iklim dingin. Gen yang relevan dengan neurobehavioral, CNTNAP2 , diidentifikasi pada populasi Chantecler dengan sinyal tertinggi ( ZHp = −3.76) dalam wilayah 340 kb. SRBD1 (protein pengikat RNA S1 1) diidentifikasi di seluruh populasi yang tinggal di lingkungan dingin. Ini telah dikaitkan kuat dengan induksi apoptosis sel (Ung et al., 2017 ). Pada populasi Tiongkok, skor ZHp terendah diamati pada Kromosom 24, yang berisi 10 gen berbeda ( BCO2 , TEX12 , IL18 , SDHD , PIH1D2 , DLAT , C11ORF52 , CRYAB , HSPB2 , dan DIXDC1 ). Gen STK38L diidentifikasi pada populasi Cina ( analisis iHS ) dan terlibat dalam pembelahan dan morfologi sel saraf (Goldstein et al., 2010 ).
Kandidat potensial dan gen untuk adaptasi stres panas membandingkan populasi dari kondisi panas-kering dan dingin
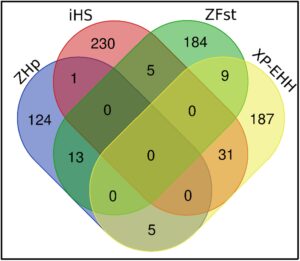
Kami menganalisis 94.610 hingga 95.035 dan 94.025 hingga 95.185 jendela di seluruh populasi dengan analisis ZF ST dan XP-EHH , masing-masing (Tabel S13 ). Untuk menyederhanakan analisis, kami hanya mempertimbangkan jendela kandidat yang tumpang tindih pada persentil ekstrem 0,001 dari distribusi empiris. Ini termasuk 392 dan 336 daerah sapuan untuk ZF ST dan XP-EHH , masing-masing, di seluruh perbandingan. Khususnya, sapuan ZF ST yang signifikan terdeteksi di seluruh autosom, kecuali pada kromosom 16, 17, 18, 22, 23, 24, 25, 26, 27, 28, 31, 32, dan 33 (Gambar 5 ). Sapuan XP-EHH diamati di seluruh autosom kecuali kromosom 3 (Gambar 6 ). Jendela-jendela ini secara kolektif mencakup 226 gen ( analisis ZF ST ) dan 275 gen ( analisis XP-EHH ) untuk sembilan perbandingan berpasangan populasi dari kondisi panas-kering (Oman, Arab Saudi, dan Fayoumi) dibandingkan dengan lingkungan dingin (Chantecler, Dulong dan Tibet) Tabel 4. Hasil terperinci disediakan dalam Tabel S14a–j dan (Gambar S3a–d dan S4a–f ) untuk ZF ST dan Tabel S15a–j dan Gambar S5a–c dan S6a–d untuk XP-EHH . Gen-gen, yang diidentifikasi menggunakan kombinasi setidaknya dua metode (Tabel 5 dan Gambar 7 ), ditemukan terkait dengan sifat adaptasi lingkungan yang berbeda, termasuk apoptosis, pengaturan tekanan darah, pengaturan pigmentasi kulit, hipoksia, pengaturan keseimbangan antara penyimpanan energi dan pengeluaran energi, metabolisme, dan respons imun. Lima istilah jalur KEGG yang terkait dengan jalur biologis yang berbeda: pematangan oosit yang dimediasi progesteron, pensinyalan adrenergik dalam kardiomiosit, jalur pensinyalan GnRH, kontraksi otot polos vaskular, dan meiosis oosit, didefinisikan untuk gen-gen ini (Tabel S16 ).